《BMC Genomics》在线发表井冈霉素生物合成温度调控的功能组学的研究成果
7月24日《BMC Genomics》在线发表了我室博士生吴杭和曲爽为共同第一作者的题为“Genomic and transcriptomic insights into the thermo-regulated biosynthesis of validamycin in Streptomyces hygroscopicus 5008”的研究论文。
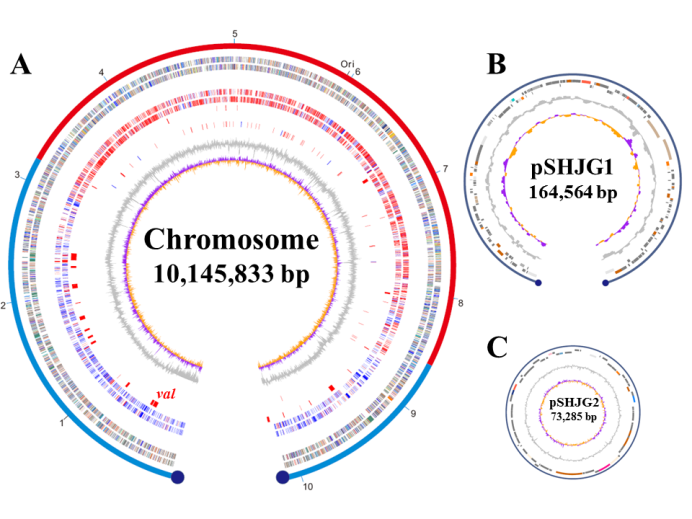
井冈霉素产生菌吸水链霉菌井冈变种5008最适生长温度为30℃左右,但是井冈霉素的产量在37℃明显提高,工业生产中的发酵温度甚至高达42℃。为了系统揭示井冈霉素高温发酵的分子基础,我们首先构建了该菌株的完整基因组图谱,比较分析发现其染色体具有较小核心区、最短末端反向重复序列(14bp),编码更多的α/β水解酶、MFS转运蛋白和逆性相关的Mg2+/Mn2+依赖的磷酸酶。
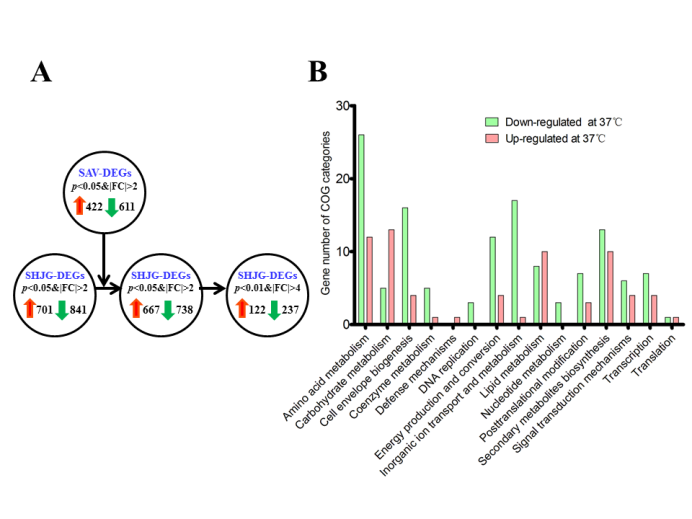
我们进一步通过芯片技术开展了该菌株在30℃和37℃不同发酵条件下的转录组分析。在37℃条件下,7.5%的蛋白编码基因的表达得到显著提高,展现出特异性的转录谱变化。首先,基因缺失发现催化谷氨酸脱氨生成α-酮戊二酸的谷氨酸脱氢酶参与了井冈霉素的合成,并证实谷氨酸是井冈霉素生物合成的直接氮来源。其次,基因缺失、回补与转录分析识别了一个链霉菌抗生素调控蛋白SARP基因负责井冈霉素生物合成的温度调控。另外,通过1个与逆性调节有关的sigma因子和2个heat-shock蛋白的基因缺失实验,发现井冈霉素生物合成与链霉菌的heat-shock调控系统紧密相关。本研究为深入解析井冈霉素生物合成的级联温度调控以及链霉菌的heat-shock响应机制奠定了新基础。
Copyright © 2016上海交通大学分子微生物学研究室
地址:上海市华山路1954号上海交通大学哲生馆(原科学馆) 电话:021-62932943
